– analiza różnorodności mikroorganizmów na powierzchni obiektów dziedzictwa kulturowego oraz w środowisku ich przechowywania
Dysponent: Uniwersytet Ekonomiczny w Krakowie
Kontakt: dr hab. Tomasz Sawoszczuk, prof. UEK ![]()
Opis metody
Metoda PCR-DGGE (ang. polymerase chain reaction – denaturing gradient gel electrophoresis) – czyli reakcja łańcuchowa polimerazy w połączeniu z elektroforezą w gradiencie czynnika denaturującego. Umożliwia ona pozyskanie informacji o strukturze genotypowej mikroorganizmów obecnych w badanym środowisku na podstawie analizy różnic w wybranych sekwencjach genomu. Umożliwia ona także, identyfikację taksonów obecnych w środowisku oraz śledzenie zmian bioróżnorodności w czasie. Największą zaletą metody PCR-DGGE jest możliwość pominięcia etapu hodowli w badaniach mikrobiologicznych, co jest niezwykle ważne, gdyż zaledwie niewielki procent mikroorganizmów można wyhodować w warunkach laboratoryjnych.
Zastosowanie
W dziedzinie badania obiektów zabytkowych metoda znajduje zastosowanie przede wszystkim do:
- identyfikacji zagrożenia mikrobiologicznego obiektów dziedzictwa kulturowego,
- badanie poziomu skażenia mikrobiologicznego obiektów i środowiska,
- identyfikacji mikroorganizmów w celu wykrycia grzybów i bakterii posiadających potencjał niszczący,
- monitorowanie czystości mikrobiologicznej powietrza wewnętrznego pomieszczeń przechowywania obiektów
Aparatura
W badaniach wykorzystywane są:
- termocykler T100 BioRad,
- zestaw do elektroforezy poziomej Polygen,
- zestaw do DGGE – DCode Universal Mutation Detection System BioRad,
- próbnik powietrza Mas-100 Eco Merck.
Warunki pomiaru
W przypadku badania obiektów próbki pobiera się w miejscu ich przechowywania, stosując sterylne wymazówki – pobór kontaktowy, nie niszczący. W przypadku analizy środowisk, próbki powietrza pobiera się próbnikiem Mas-100 Eco w badanym pomieszczeniu.
Format wyników
Przedstawiona procedura badawcza umożliwia przeprowadzenie analizy ilościowej genotypów obecnych w badanym środowisku, a także ocenę jakościową dominujących taksonów mikroorganizmów.
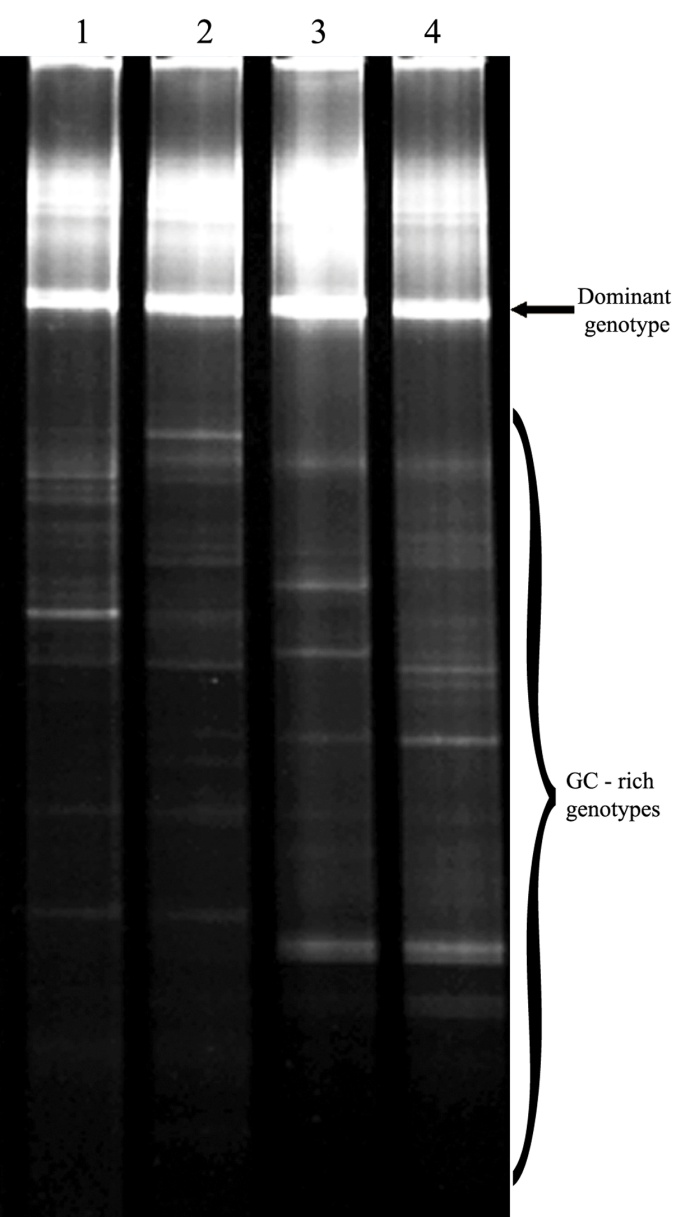 Rys. 1. Przykładowy fingerprint otrzymany dla próbek powietrza (Lech et al. 2015). |
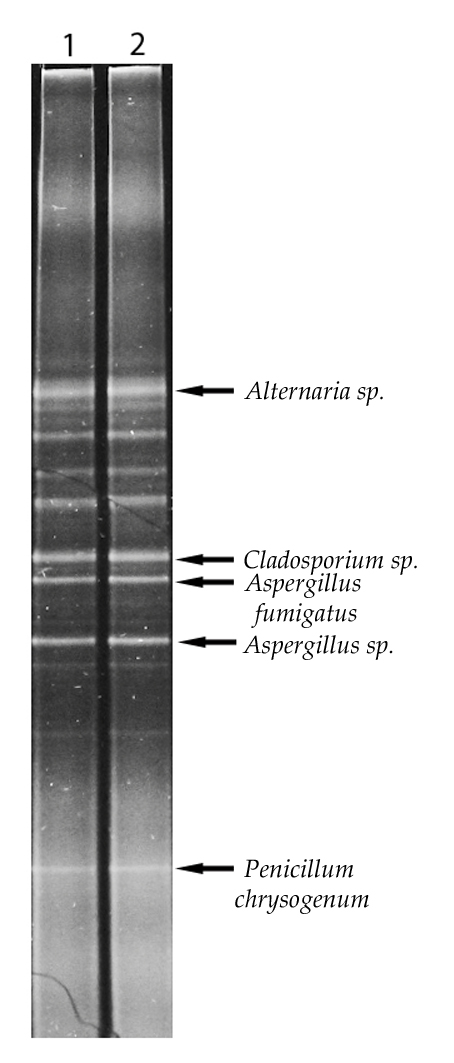 Rys. 2. Przykładowy fingerprint dla powierzchni dokumentu pergaminowego (Lech, 2016). |
Bibliografia
Lech Tomasz, 2016, Evaluation of a Parchment Document, the 13th Century Incorporation Charter for the City of Krakow, Poland, for Microbial Hazards, Applied and Environmental microbiology 82:9, doi:10.1128/AEM.03851-15.
Lech Tomasz, Ziembinska-Buczynska Aleksandra, 2015, Evaluation of a Modified Sampling Method for Molecular Analysis of Air Microflora, Genetics and Molecular Research, 14 (2), 3200-3208.

